案例简介
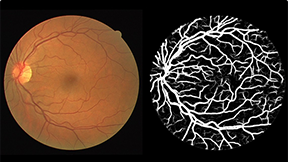
视网膜眼底图像分析可以间接反应脑血管系统变化。视网膜血管是视网膜中央动脉和视网膜中央静脉的终末支,供应大脑前三分之二区域的大脑前、中动脉和视网膜动脉均起源于颈内动脉。视网膜血管具有与脑血管相似的解剖学、生理学和胚胎学特征,视网膜血管的变化反映了脑血管的类似变化。因此,研究视网膜血管变化可以为了解脑卒中及相关脑血管疾病的病理生理学提供线索。利用视网膜血管系统作为大脑血管系统状态的标志具有明显的优势,视网膜血管是唯一可使用视网膜成像技术(如眼底照相机)直接观察到身体里的血管,具有非侵入式的优点。近年来,视网膜眼底图像分析技术的应用已经越来越普遍,并且提供了越来越复杂的技术来分析视网膜血管形态学的不同方面,例如视网膜微血管的宽度、血管跟踪等。这些技术的自动化程度较低且客观,为研究视网膜微血管异常作为脑血管病理学标志的潜力提供了机会。图是眼底图像及血管分割标注。如果计算机可以根据眼底图像预测出血管的结构,则可对眼科医生提供帮助。
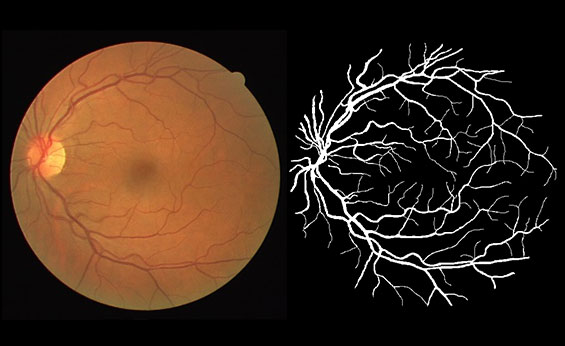
眼底图像(左)与血管标注(右)
近些年,深度学习技术在图像分类、物体检测、语音识别等处理领域取得了瞩目的成就。深度神经网络作为一种非线性的特征提取及分类方法已被广泛应用于医学影像分析。因此,有大量研究者利用深度学习技术对眼底血管进行分割。但是,在实际应用中,由于血管分割模型存在参数量大、计算时间复杂度高等问题,需要昂贵的GPU才能达到较快的分割速度。这无疑增加了模型部署的成本,限制了其使用范围,并使得模型难以应用在小型移动终端上(比如智能手机、手持式医疗设备)。因此,本项目是开发一套从眼底图像采集到输出血管分割结果的系统。其中,利用Atlas 200DK的神经网络计算能力,提高模型运行效率,来实时的对眼底血管进行分割。
本案例完成的系统在华为Atlas 200DK上实现了对眼底图像的血管分割,具有完整性、代表性和实用性,满足了在实际场景下用对眼底血管进行分割的实时性需求。
系统总体设计
该系统包括眼底图像采集子系统、用户交互子系统和模型处理子系统。图像采集子系统通过眼底相机拍摄眼底图像,并将眼底图像保存到硬盘中。用户交互子系统负责界面展示、与Atlas 200DK的通信等。模型处理子系统复杂在Atlas 200DK上运行血管分割模型。
用户交互子系统将本地硬盘上眼底图像传送到Atlas 200DK,并启动Atlas上的血管分割模型,之后,将血管分割结果拷贝到本地,并展示分割结果图。
功能结构
眼底视网膜血管图像分割系统可以划分为图像采集及处理、模型构建、用户交互等三个主要子系统。其中图像采集及处理包括图像采集、图像尺寸归一化和图像保存等;模型构建包括网络定义、模型训练和模型推理等;用户交互包括用户选择本地眼底图像、启动分割模型、展示分割结果等公能。为了说明各模块之间的结构关系,细化的系统整体功能结构图如图所示。
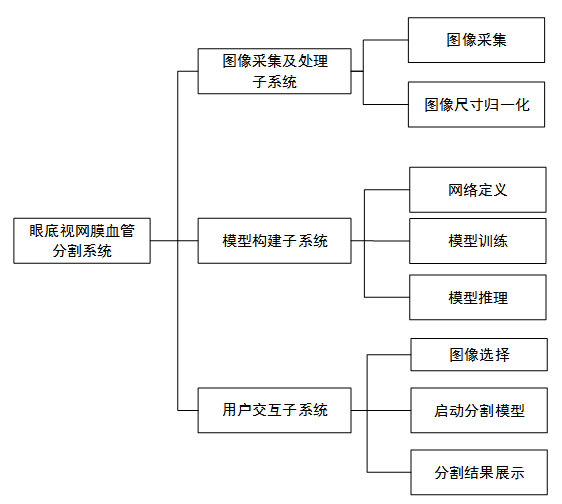
系统整体功能结构图
运行流畅与体系结构
按照体系结构划分,整个系统的体系结构可以划分为三部分,分别为主机端的训练层、Atlas 200DK端的推断层以及本地端的用户交互层,如图所示。各层侧重点各不相同。训练层运行在安装有Caffe框架和NVIDIA GPU加速卡的工作站。推断层运行于Atlas 200DK环境,能够支持卷积神经网络的加速。用户交互层运行于安装有Python环境的本地端,能够与用户及Atlas 200交互并实时显示推断层的计算结果。各层之间存在依赖关系。推断层需要的血管分割网络由训练层提供。相应的,用户交互层要显示的数据需要由推断层计算得到。
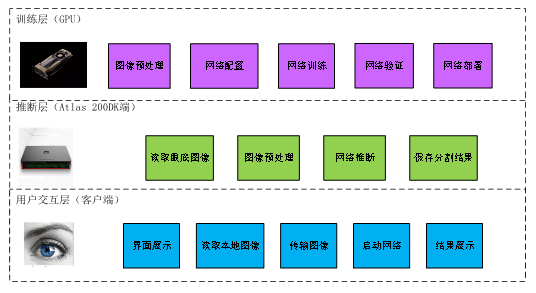
系统的体系结构
系统设计与实现
本节详细介绍系统的设计与实现。“数据集制作”给出眼底图像训练集的制作;'网络训练'节介绍如何基于Caffe训练一个血管分割网络;“模型部署”解释如何借助MindStudio将模型部署到Atlas 200DK上,并执行模型的推理并保存分割结果。最后“用户交互层”介绍本地客户端的设计与实现。
数据集制作
使用公开的眼底血管分割数据集DRIVE [1]。DRIVE是目前使用最为广泛的评估眼底血管分割模型的公开数据集。它共包含40张分辨率为565*584的彩色眼底图像。训练集包括20张图像,测试集包括20张图像。训练集中的每张图片都提供了一组专家的标注结果及mask文件。测试集中的每张图片都提供了两组专家的标注结果及mask文件。第一组专家的标注结果作为金标准,用来评估血管分割模型,第二组专家的标注用来与计算机算法进行对比。眼底图像以tif格式存储,标注结果和mask文件以gif格式存储。测试集上的眼底图像、标注和mask文件如图所示。
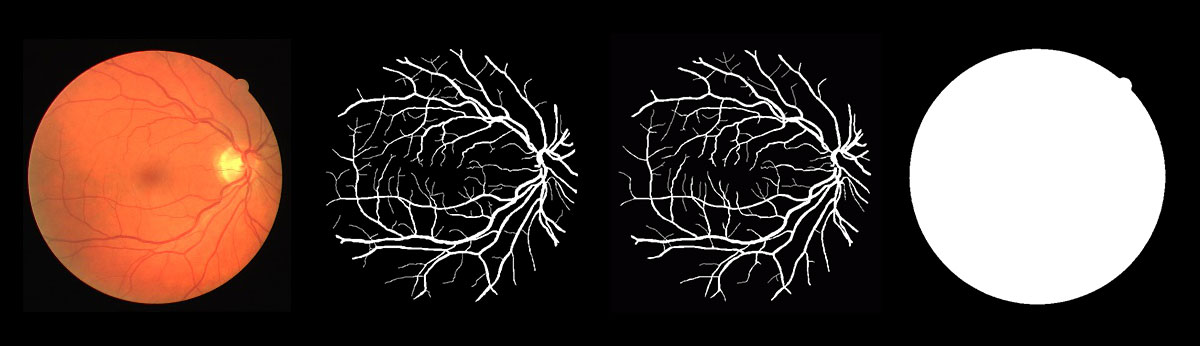
RIVE测试集图片(从左到右:眼底图像、第一专家标注、第二专家标注、mask)
使用训练集中的20张眼底图像训练血管分割模型。首先对眼底图像进行预处理。这里,使用matlab将图像及专家标注尺寸缩放到512*512,如清单所示。
清单0-1 resize_drive.m脚本
Path_img = "./img/";
Path_la = "./label/";
Img_all = dir([Path_img "*.gif"]);
for i=1:length(Img_all)
im = imread([Path_img Img_all(i).name]);
la = imread([Path_la Img_all(i).name(1:2) "_manual1.gif"]);
im = imresize(im, [512 512]);
la = la>1;
la = imresize(la, [512 512]);
imwrite(im, [Path_img Img_all(i).name(1:end-4) ".jpg"]);
imwrite(uint8(la), [Path_la Img_all(i).name(1:2) "_manual1.tif"]);
end图像缩放到512*512之后,眼底图像以jpg格式存储,专家标注以tif格式存储,血管像素标注为1,其余像素标注为0。然后,使用旋转和镜像对训练集进行扩充。具体包括旋转90、180、270度和垂直翻转与水平翻转。扩充之后训练集图像共包括120张眼底图像。同样,扩充操作使用matlab进行。具体如清单所示。
清单0-2 aug_drive.m 旋转90度
angel = 90;
Path_img = "./img";
mkdir(strcat(Path_img,'_',num2str(angel)));
img_all = dir(strcat(Path_img,'/*.tif'));
for i=1:length(img_all)
im=imread(strcat(Path_img, '/', img_all(i).name));
im=imrotate(im, angel);
%im=flip(im,1);im=flip(im,2);%翻转
imwrite(im, strcat(Path_img,'_',num2str(angel),'/', img_all(i).name));
end网络训练
使用自定义的全卷积神经网络进行血管分割任务。网络结构如图所示。网络一共包括16个卷积层,conv1到conv12每个卷积核大小为3×3,stride为1,厚度为16。conv13到conv15每个卷积核大小为3×3,stride为1,厚度为8。conv16使用3×3卷积核得到单通道的特征图。随后,经过sigmoid得到血管分割概率图。
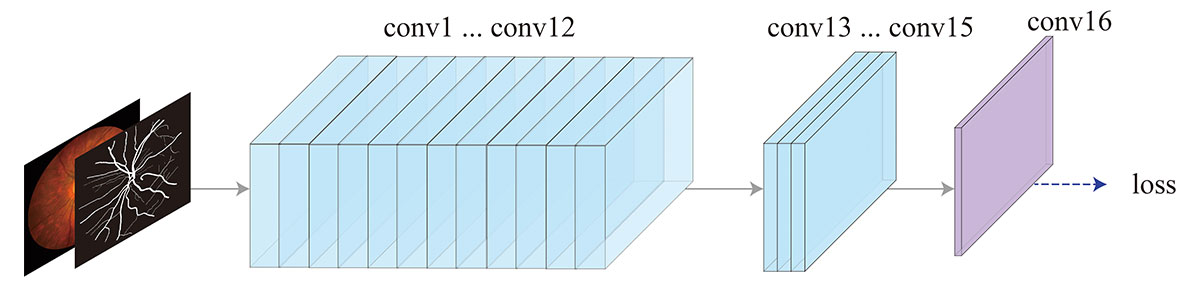
血管分割网络结构图
Caffe的网络定义主要在prototxt中完成,其网络的数据层(仅给出训练集的定义,测试集可以类似定义)如清单所示。其中,ImageSegData层并未官方Caffe自带的层,可从https://github.com/guomugong/FFIA下载编译。mirror:true意味着对每一张图片会进行随机镜像,crop_size的意思是对每一张眼底图像随机剪裁出500*500的图像块进行前向传播。
清单0-3 网络数据层定义
# train phase
layer {
name: "data"
type: "ImageSegData"
top: "data"
top: "label"
transform_param {
mirror: true
crop_size: 500
}
image_data_param {
root_folder: ""
source: "train_drive_512.lst"
batch_size: 1
shuffle: false
label_type: PIXEL
}
}train_drive_512.lst保存图像和标注路径。如清单所示,共包括120行,每行代表图像-标注对。在shuffle设置为false时,ImageSegData层逐行读取此文件,将图像和标注读取到内存。
清单0-4 标签文件
512/img/21_training.jpg 512/label/21_manual1.tif
...
512/img/40_training.jpg 512/label/40_manual1.tif
512/img_90/21_training.jpg 512/label_90/21_manual1.tif
...
512/img_90/40_training.jpg 512/label_90/40_manual1.tif
512/img_180/21_training.jpg 512/label_180/21_manual1.tif
...
512/img_180/40_training.jpg 512/label_180/40_manual1.tif
512/img_270/21_training.jpg 512/label_270/21_manual1.tif
...
512/img_270/40_training.jpg 512/label_270/40_manual1.tif
512/img_m1/21_training.jpg 512/label_m1/21_manual1.tif
...
512/img_m1/40_training.jpg 512/label_m1/40_manual1.tif
512/img_m2/21_training.jpg 512/label_m2/21_manual1.tif
...
512/img_m2/40_training.jpg 512/label_m2/40_manual1.tif网络输出层定义如清单所示。
清单0-5 网络输出层定义
layer {
name: "predict"
bottom: "conv1_15"
top: "score"
type: "Convolution"
param {
lr_mult: 1
decay_mult: 1
}
param {
lr_mult: 2
decay_mult: 0
}
convolution_param {
num_output: 1
pad: 1
kernel_size: 3
weight_filler {
type: "xavier"
}
}
}
# 训练阶段
layer {
name: "loss"
bottom: "score"
bottom: "label"
top: "loss"
type: "SigmoidWeightedCrossEntropyLoss"
}
# 测试阶段
layer {
name: "output"
bottom: "score"
top: "output"
type: "Sigmoid"
}在训练时,由于眼底图像中血管像素比例只占10%左右,血管像素与背景像素存在类别不均衡的问题,因此,使用类平衡的交叉熵损失函数,定义如下:
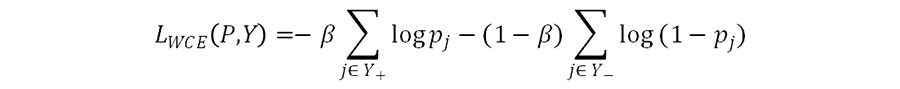
其中,
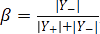
为权重因子,为血管像素集合,为背景像素集合,Pj为第j个像素经过sigmoid操作之后的激活值。
在测试时,直接对score进行sigmoid操作得到分割概率图,图中每个像素值在0和1之间。
写好网络prototxt文件后,还需在solver.prototxt中定义训练的各种参数,如清单所示,这里我们使用GPU进行计算。
清单0-6 solver.prototxt的参数定义
#网络模型prototxt路径
net: "vel_ascend.prototxt"
# 训练时不进行测试
test_iter: 0
test_interval: 10000000
display: 100
# 学习率和求解器
base_lr: 1e-3
solver_type: "Adam"
momentum: 0.90
weight_decay: 0.0005
# 最大迭代次数
max_iter: 10000
snapshot: 10000 # 快照
snapshot_prefix: "snapshot/ascend"
solver_mode: GPU最后,在命令行里启动Caffe训练程序开始训练,如清单所示。其中$CAFFE_ROOT为Caffe的根目录。
清单0-7 启动训练程序进行模型训练
$CAFFE_ROOT/build/tools/caffe train –solver solver.prototxt –gpu 0训练过程如图所示。
训练过程展示
网络训练结束之后,会生成权重文件ascend_iter_10000.caffemodel。
模型部署
要将训练好的Caffe模型应用到Atlas200 DK上,首先要将其转换为Ascend AI处理器支持的离线模型,这里需要安装MindStudio实现模型转换。MindStudio的安装主要是按照https://www.huawei.com/minisite/ascend/cn/file.html中的教程,这里不再赘述。模型部署主要包括如下步骤:
新建项目
在MindStudio中新建一个项目,Target选择Atlas DK。
添加数据集
将DRIVE的测试集添加到项目中,测试集的图像与训练集图像的预处理方式相同,都是先缩放到512*512的尺寸,以jpg的格式保存。在MindStudio的操作如图所示。数据类型Data Type选择Image,Data Source是本地的文件夹,Folder是本地存放DRIVE测试集的路径。这里选中Include YUV420SP,因为在模型转换的过程中也是使用的YUV格式。
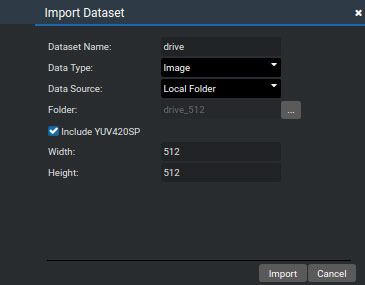
自定义添加数据集
模型转换的参数如图所示。Model Type为框架类型,这里选择Caffe,Model File为记录网络结构的prototxt文件,Weight File为模型权重文件(caffemodel)。推理过程的batch size设为1,图像的宽、高和通道数不变,因此输入的维度N、C、H、W分别对应为1、3、512、512。为了使输入图像的格式符合模型输入要求,还需在模型转换中设置图像预处理的参数,其中输入图像的格式Input Image Format需调整为YUV420SP_U8,模型图像的格式Model Image Format为BGR888_U8。 其余选项设置为off。模型转换成功之后会生成一个om文件。
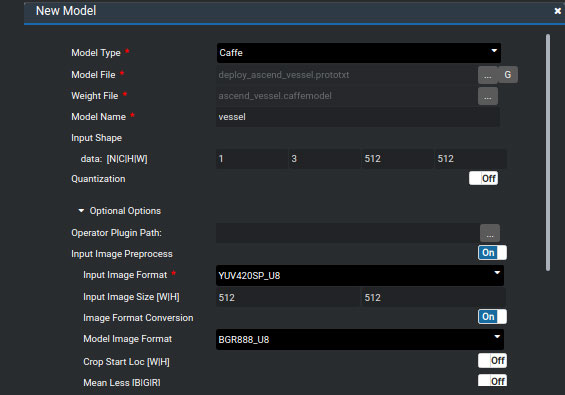
模型转换时的关键配置
模型编排
如图所示结构编排网络,其中drive为自定义添加的数据集,vessel为转换后的血管分割模型,ImagePreProcess_1为图像预处理模块,负责将图像缩放到512*512,MindInferenceEngine_1负责分割模型的推理,SaveFilePostProcess_1保存分割结果。
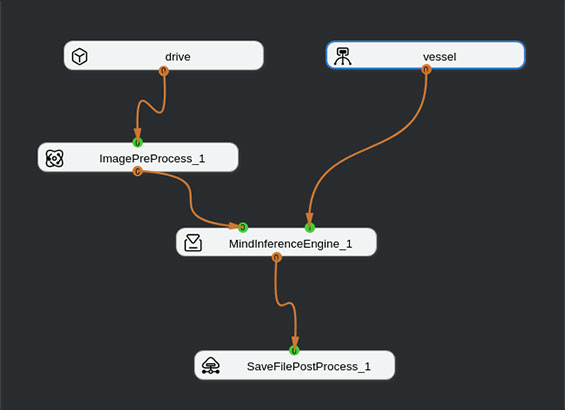
网络编排结构图
模型运行
网络编排完成之后,依次点击Save,Generate和Run。运行成功的话,分割结果会保存在out目录下。如图所示为DRIVE测试集上20张眼底图像的分割结果。每张图像的分割结果单独保存到一个文件中。
分割结构
另外,在保存分割结果时,为了提高IO效率,减少运行时间。对SaveFilePostProcess_1操作进行了修改,将分割结果以二进制的形式保存,而不是文本文件的形式,代码如清单所示。
清单0-8 SaveFilePostProcess_1以二进制形式保存分割结果
FILE* fd;
fd=fopen(outFileName.c_str(),"wb"); //打开文件
if(fd == NULL){
HIAI_ENGINE_LOG(HIAI_IDE_ERROR, "[SaveFilePostProcess_1] open file %s error!", outFileName.c_str());
delete[] result;
result = NULL;
return HIAI_ERROR;
}
int oneResultSize = size / tran->b_info.max_batch_size;
ret = fwrite((const char*)result,sizeof(float), sizeof(float) * oneResultSize,fd); //以二进制形式写入文件
if(ret == 0){
HIAI_ENGINE_LOG(HIAI_IDE_ERROR, "[SaveFilePostProcess_1] write file error!");
ret = fclose(fd);
if(ret == EOF){
HIAI_ENGINE_LOG(HIAI_IDE_ERROR, "[SaveFilePostProcess_1] close file error!");
}
delete[] result;
result = NULL;
return HIAI_ERROR;
}
ret = fclose(fd);
if(ret == EOF){
HIAI_ENGINE_LOG(HIAI_IDE_ERROR, "[SaveFilePostProcess_1] close file error!");
delete[] result;
result = NULL;
return HIAI_ERROR;
}可视化分割结果
由于分割结果以二进制的形式保存到文件中,无法直接查看,可用如清单所示matlab脚本将对应二进制文件转换成png图片。转换之后二进制文件davinci_20_test_output_0__0.txt对应的分割结果以20_test.png的名字保存。
清单0-9 convert_binfile_to_png.m将二进制文件转换成png
% 切换到二进制文件所在目录
Binfile_all = dir("./*.txt");
for i=1:length(Binfile_all)
binfile_name = Binfile_all(i).name; %获得二进制文件名
fin = fopen(binfile_name, "rb");
[im, num] = fread(fin, [512 512], "float"); %读取二进制文件
im=im';
im = imresize(im, [584 565]); %恢复图片尺寸
imwrite(im, [Binfile_all(i).name(9:15) '.png']); %保存分割图片
end用户交互层
用户交互子系统以GUI的形式与用户交互,主要功能包括与Atlas 200通信,将本地硬盘上的眼底图像传送到Atlas 200DK,并启动Atlas上的血管分割模型,最后将血管分割结果拷贝到本地,并展示分割结果图。
我们在windows平台下基于Python3开发此子系统,主要使用了tkinter,PIL,paramiko和numpy等库。此软件运行时,需要通过网线与Atlas连接,并正确设置本地IP地址。可有使用ping 192.168.1.2检查设置是否正确。
子系统主界面如图所示。由于眼底图像的采集需要专业的眼底相机,这里忽略了眼底图像采集的功能,仅模拟从本地硬盘选择眼底图像的功能。界面中的Select按钮用于选择眼底图像,Process操作执行之后开始进行血管分割。

用户交互子系统界面
主界面显示代码如清单所示。
清单0-10 界面显示
import tkinter as tk
app = tk.Tk()
app.title('Vessels')
app.geometry('560x630')
Frame=tk.LabelFrame(app,padx=10,pady=10)
Frame.place(x=10,y=15)
cover=Image.open('./cover.jpg')
coverimg=ImageTk.PhotoImage(cover)
lbPic = tk.Label(Frame,image=coverimg)
lbPic.grid(row=0,column=0)
Frame_button=tk.LabelFrame(app,padx=10,pady=10)
Frame_button.place(x=295,y=565)
Button_file=tk.Button(Frame_button,text='Select',width=10,relief=GROOVE,fg='#5D88B5',command=choosepic)
Button_file.grid(row=0,column=0)
Button_run=tk.Button(Frame_button,text='Process',width=10,relief=GROOVE,fg='#71195C',command=process)
Button_run.grid(row=0,column=2)
app.mainloop()用户点击select从本地选择眼底图像,如图所示,此部分代码如清单所示。
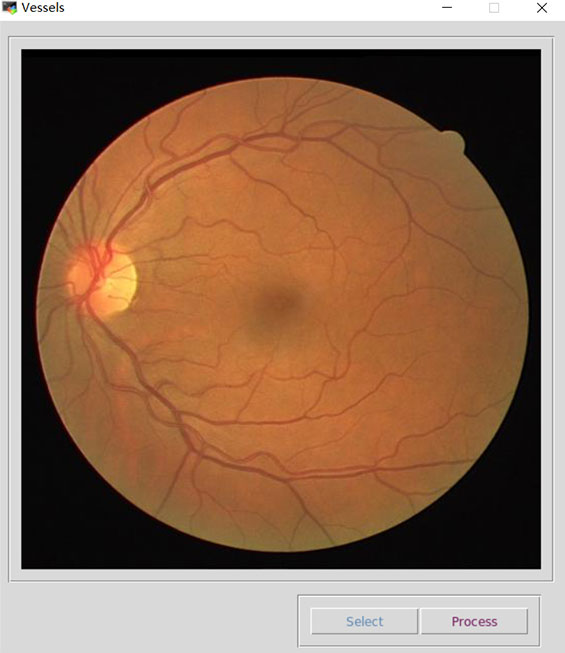
选择本地图片
清单0-11 选择本地图片
def choosepic():
lobal filename
filename=askopenfilename()
if filename != '':
im=Image.open(filename)
img=ImageTk.PhotoImage(im)
lbPic.config(image=img)
lbPic.image = img图片选中之后,用户可点击Process进行下一步操作。Process操作对应着与Atlas通过ssh进行连接,将用户选中的眼底图像放到Atlas的指定目录下,运行Atlas上shell脚本,最后从Atlas的指定目录取回血管分割文件并展示。系统基于paramiko库使用ssh与Atlas通信的操作如代码清单所示。
清单0-12 连接Atlas
ip_addr='192.168.1.2'
port=22
username='HwHiAiUser'
password=''
filepath='/home/HwHiAiUser/xxxx/test.jpg' #Atlas存放眼底图像路径
scp=paramiko.Transport((ip_addr,port))
scp.connect(username=username,password=password)
sftp=paramiko.SFTPClient.from_transport(scp)
sftp.put(filename,filepath) #将用户选择的眼底图像拷贝到Atlas指定目录下
...
ssh.exec_command(cmd) #Atlas上执行cmd脚本,启动分割进程
sftp.get('/home/HwHiAiUser/HIAI_PROJECTS/workspace_mind_studio/vessels/out/result_files/0/SaveFilePostProcess_1/xxx.txt',resultfile) #取回分割结果正如在前几节所介绍,分割结果以二进制形式保存到txt文件中,无法直接显示,因此,需要将其转换成图片的形式,一方面展示给用户,另一方面保存到本地,方便后续查看。在Python下,实现二进制文件转换成图片的代码如清单所示。
清单0-13 二进制文件转图片 - python
import os
import struct
import numpy as np
import matplotlib as plt
filename='test.txt'
result=[]
with open(filename, "rb") as fin:
while True:
data=fin.read(4)
if not data:
break
elem = struct.unpack("f", data)[0]
result.append(elem)
fin.close()
pic=np.array(result)
pic=np.reshape(pic*255,(512,512))
plt.pyplot.imshow(pic)案例系统部署
案例系统最终需要部署模型推理层和用户交互层,推理层部署到Atlas 200平台上,负责眼底图像的分割。用户交互层对用的系统部署到配有相关开发环境可以与用户交互的台式机或者笔记本上。台式机与Atlas通过网线连接。需要注意的是,仅仅使用Atlas 200的话也是可以实现对眼底图像的分割,但是只能处理开发版上的眼底图像。加入用户交互层的话,则可以实现从眼底图像采集,眼底血管分割,保存分割结果等完整的操作流程。部署流程图如图所示。
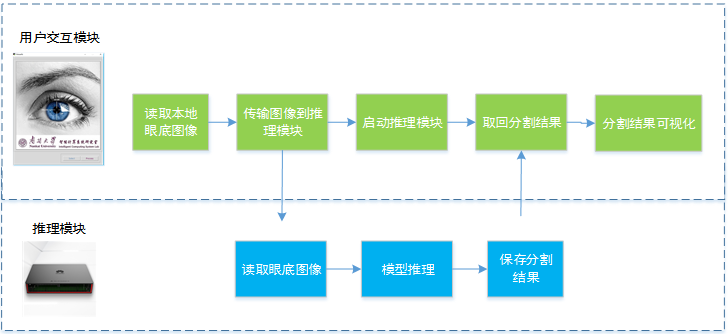
部署流程图
部署流程图中各个模块的作用在前几节已详细论述,这里不在阐述。部署完成之后,在真实场景下,应如图所示。Atlas 200DK作为神经网络加速器,提高血管分割模型运行效率,用户交互层作为主控程序与Atlas 200与用户交互。

真是场景部署图
运行结果
我们在DRIVE数据集上对血管分割模型进行训练和测试。图为测试集上的ROC曲线,AUC值为0.9646。图为我们从20张测试眼底图像中选中的四张图像的血管分割结果。
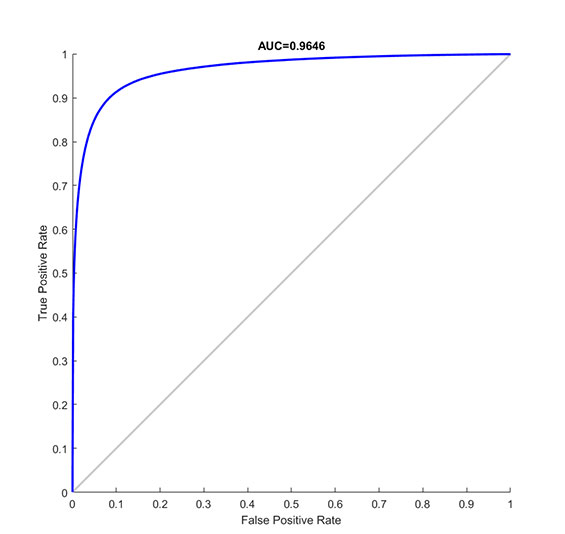
ROC曲线
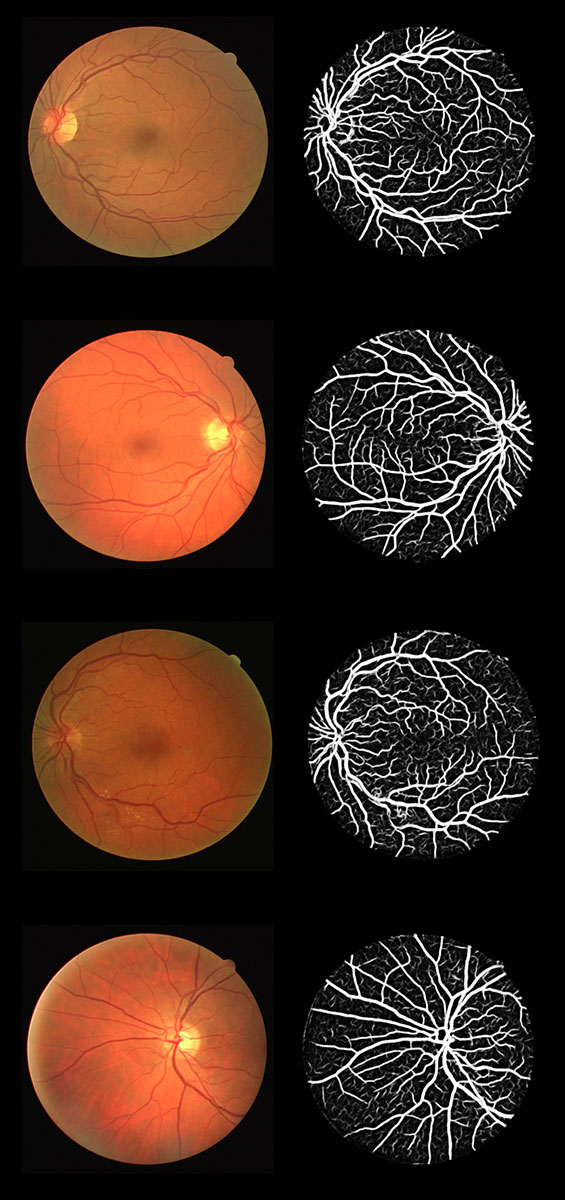
血管分割结果(左图为眼底图像,右图为分割概率图)
我们使用matlab脚本计算AUC值,代码如清单所示,需要注意的是计算AUC的脚本需要用到DRIVE数据集的标注和mask文件。
清单0-14 compute_roc.m
path='./分割结果/'; %保存分割结果的路径
test_all=dir([ path '*.txt']);
num_test_images=length(test_all);
label = zeros(1,584*565*num_test_images);
pred = zeros(1,584*565*num_test_images);
ii = 1;
for i=1:num_test_images
image_filename=[path test_all(i).name];
label_filename=['./DRIVE_DATASET/label/' test_all(i).name(9:10) '_manual1.gif'];
mask_filename=['./DRIVE_DATASET/mask/' test_all(i).name(9:15) '_mask.gif'];
la = imread(label_filename);
ma = imread(mask_filename);
fin=fopen(image_filename,'rb');
[im,num]=fread(fin,[512,512],'float');
im=im';
im = imresize(im,[584 565]); %需要resize到原图尺寸
im = double(im);
for j=1:size(im,1)
for k=1:size(im,2)
if ma(j,k) == 255
if la(j,k) == 0
label(1,ii) = 0;
else
label(1,ii) = 1;
end
pred(1,ii) = im(j,k);
ii = ii+1;
end
end
end
end
fclose(fin);
label=label(1,1:ii-1);
pred=pred(1,1:ii-1);
[x y t auc op]=perfcurve(label,pred,1);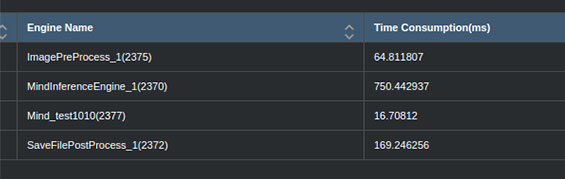
性能分析
最后,我们测算了系统的主要时间消耗情况。20张尺寸为512*512的眼底图像的总耗时(图片预处理时间+推理时间+保存结果时间)约为1000ms。20张图像的推理时间为750ms,平均一张图像的推理耗时37.5ms。
本章小节
本案例属于典型的基于深度学习的医疗影像分析任务。本章提供了一个基于华为Atlas 200DK的眼底视网膜血管图像分割案例。该案例演示了如何利用交互层与Atlas 200DK的神经网络计算能力实现实时地眼底图像的血管分割。
本文围绕如何在Atlas 200DK端部署血管分割模型做了详细的介绍,包括系统的整体结构、功能和运行流程,并详细介绍了图像采集及预处理、模型构建和用户交互这三个子系统。讲述了如何使用DRIVE数据集在Caffe框架下训练血管分割网络,如何将分割网络部署到Atlas 200DK端,用户交互层如何利用Atlas执行血管分割操作等。部署后的系统在DRIVE数据集上进行测试,结果表明案例系统具有较快的推理速度和较好的分割结果。
参考资料
[1] J. Staal, M. Abrmoff, M. Niemeijer, M. Viergever, and B. van Ginneken, “Ridge-based vessel segmentation in color images of the retina,” IEEE TMI, vol. 23, no. 4, pp. 501–509, 2004.
[2] https://www.huawei.com/minisite/ascend/cn/file.html
[3] https://github.com/guomugong/FFIA